COVID-19
Depuis l'identification initiale du nouveau coronavirus SARS-CoV-2 comme agent causal du COVID-19, près de 15 millions d'infections (au 23 juillet 2020) ont été enregistrées dans le monde entier. Grâce aux efforts de recherche mondiaux, une image de la biologie du virus et de ses effets sur l'hôte humain est en train d'émerger. Nous offrons une grande variété de produits de haute qualité pour la recherche sur COVID-19.
Anticorps, kits, protéines par phase de la maladie:
Produits COVID-19 par type de produit:
Infection SARS-CoV-2
L'infection par le SARS-CoV-2 est principalement déclenchée lorsque le récepteur de surface de l'enzyme de conversion de l'angiotensine 2 (ACE2) de la cellule hôte est lié par la protéine du pic (S) du virus par l'intermédiaire de son domaine de liaison au récepteur (RBD). L'ECA2 est codée sur le chromosome X, ce qui pourrait expliquer le taux de mortalité plus élevé de la COVID-19 chez les hommes. Il est possible que le fait d'avoir deux allèles différents de l'ECA2 confère un certain degré de résistance.
La liaison du SARS-CoV-2 à l'ECA2 déclenche l'amorçage de la protéine trimère S au site de clivage polybasique S1/S2 par la protéase transmembranaire sérine 2 associée à la surface cellulaire (TMPRSS2) et, à un moindre degré, par la cathepsine B et L. L'ectodomaine S1 contenant la RBD détermine le tropisme cellulaire et la fixation du virus à sa cellule cible. L'endodomaine S2 héberge un domaine transmembranaire et est impliqué dans l'entrée du virus par endocytose. Il contient également un second site de protéase, le site de clivage S2 de type furine. Le préclivage de ce site de clivage de type furine pourrait expliquer la plus grande infectiosité du SARS-CoV-2 par rapport au SARS-CoV (qui ne possède pas ce site).
Produits liés à la COVID-19:
- (8)
- (15)
- (1)
- (4)
- (5)
- (9)
- (8)
- (5)
- (3)
- (4)
- (3)
- (1)
Réplication virale
À l'entrée, le génome de l'ARNs(+) viral de sens positif est libéré. Deux grands cadres de lecture ouverts polycistroniques ORF1a et ORF1b à l'extrémité 5' du génome codent pour 16 protéines non structurelles (NSP) formant deux polyprotéines réplicases pp1a et pp1b. Nsp3 contient un domaine de protéase de type papaïne (PLpro) et traite Nsp1-4 de pp1a. La principale protéase de type chymotrypsine 3C (Mpro, 3CLpro, Nsp5) du SARS-CoV-2 digère les sites de clivage protéolytique restants.
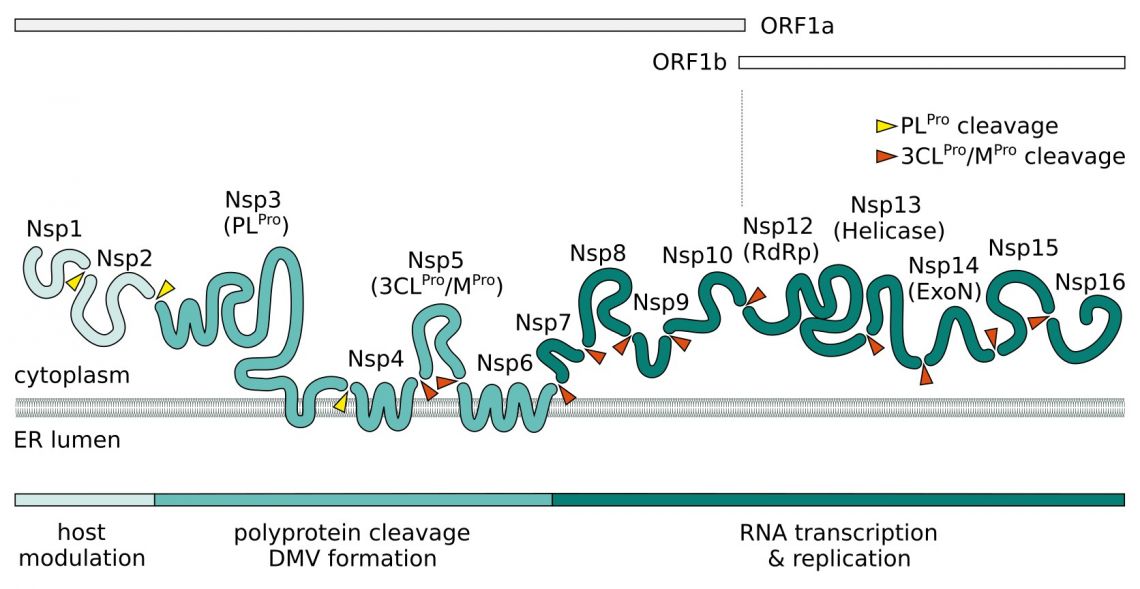
On pense que Nsp1 et Nsp2 jouent un rôle dans la modulation de l'hôte pour supprimer une réponse antivirale. Un complexe constitué des protéines transmembranaires Nsp3, Nsp4 et Nsp6 induit la formation de vésicules à double membrane (DMV) améliorant la réplication virale par le biais de la réplication associée à la membrane et affectant l'autophagie. En plus de ses PSN, SARS-CoV-2 recrute des protéines hôtes pour former un complexe de réplication et de transcription (RTC). Le composant central pour la réplication de l'ARNs(+) est l'ARN polymérase ARN-dépendante (RdRp). Elle forme le complexe de réplication avec Nsp7 et Nsp8. Celles-ci servent de primase et génèrent de courtes amorces d'ARN pour la RdRp dépendante de l'amorce et augmentent sa capacité de traitement. Nsp9 a une préférence pour l'ARNs et on pense qu'il interagit avec Nsp8 dans le complexe de réplication. Nsp13 et Nsp16/Nsp10 ont respectivement une activité hélicase/triphosphatase et méthyltransférase et coiffent l'ARNm viral naissant. L'exonucléase Nsp14 (ExoN) dote le mécanisme de réplication d'une fonction de correction, augmentant ainsi la fidélité de la synthèse de l'ARN du SARS-CoV-2. La dernière protéine du complexe de réplication est l'endoribonucléase Nsp15 (EndoU), spécifique de l'uridine.
Une fois que le RTC est assemblé, un ARNd intermédiaire est synthétisé à partir de l'ARNs(+) génomique. Cet intermédiaire sert de modèle pour la production d'ARN sous-génomique (sgRNA) et de nouveaux ARNs(+) génomiques complets. Les premiers sont transcrits dans les quatre protéines structurelles du virus (N, E, M et S) et les neuf protéines accessoires codées dans la section 3' du génome. Le second est encapsulé dans les phosphoprotéines de la nucléocapside (protéine N) puis enveloppé par la protéine d'enveloppe (protéine E), la glycoprotéine membranaire (protéine M) et la protéine de pointe (protéine S) pour former de nouveaux virions.
Produits d'anticorps liés à la COVID-19:
- (8)
- (15)
- (1)
- (9)
- (8)
- (1)
- (2)
- (3)
Produits protéiques liés à la COVID-19:
- (9)
- (6)
- (1)
- (2)
COVID-19
La gravité de la maladie chez les patients est due non seulement à l'infection virale mais aussi à la réponse de l'hôte. La réponse inflammatoire de l'hôte influence fortement la détérioration des voies respiratoires. Dans 70 % des cas de COVID-19 mortels, les syndromes de détresse respiratoire aiguë (SDRA) qui en résultent conduisent directement à une insuffisance respiratoire. La deuxième cause la plus fréquente de décès dans le contexte de la COVID-19 est une réponse inflammatoire systémique incontrôlée provoquée par une surproduction de marqueurs inflammatoires. Ce syndrome dit de libération de cytokines ou "tempête de cytokines" est un facteur important du SDRA et du syndrome de dysfonctionnement de plusieurs organes (SDMO), qui cause des dommages notamment au cœur, aux reins et au foie.
SARS-CoV-2 Infection et réponse inflammatoire
Le SARS-CoV-2 se réplique principalement dans les voies respiratoires inférieures où il provoque une pneumonie et un SDRA. Alors que les protéines structurelles et non structurelles du virus sont principalement chargées de la construction du virion et de la réplication du virus respectivement, au moins certains des membres du troisième groupe de protéines SARS-CoV-2, les neuf facteurs accessoires (Orf3a-10), ont été impliqués dans la progression de COVID-19.
SARS-CoV-2 L'Orf3a induit l'apoptose dans des modèles de lignées cellulaires et on pense qu'il active le NF-kB et l'inflammasome NLRP3 impliqué dans la pyroptose, une forme d'apoptose hautement inflammatoire. Il a été démontré que l'Orf8b induit un stress du RE et active également l'inflammasome NLRP3. Cela suggère que l'infection virale des cellules épithéliales des voies respiratoires conduit à la pyroptose, une forme hautement inflammatoire de mort cellulaire programmée. La pyroptose s'accompagne généralement de la libération de cytokines pro-inflammatoires, ce qui entraîne le recrutement de cellules immunitaires supplémentaires et une amplification supplémentaire de la réponse immunitaire. L'Orf3a induit également la sécrétion du marqueur de pyroptose IL-1 bêta. L'Orf7a peut également jouer un rôle dans la pathogénèse par son rôle dans l'apoptose induite par le virus.
L'étendue des dommages aux tissus des voies respiratoires inférieures peut être surveillée dans les premiers stades de la COVID-19 en utilisant les niveaux de protéine C-réactive (PCR) comme indicateur de la gravité de la maladie.
La destruction des cellules épithéliales alvéolaires libère les motifs moléculaires associés aux dommages (DAMP) et les motifs moléculaires associés aux agents pathogènes (PAMP), qui sont détectés par les récepteurs de reconnaissance de motifs (PRR) sur les cellules épithéliales alvéolaires et les macrophages. Les principaux PRR pour les ARN viraux sont des membres de la famille des récepteurs RIG-I-like (RLR). En se liant à l'ARN viral, un changement de conformation des RLR déclenche l'agrégation des MAVS et la formation du signalosome des MAVS. Le signalosome MAVS déclenche la dimérisation de l'IRF3/7 et active la voie NF-κB, conduisant à la production d'IFN de type I, les cytokines antivirales les plus importantes, et de cytokines pro-inflammatoires IL-6, IFN-gamma, CD46 et CXCL10. La sécrétion coordonnée de cytokines et de chimiokines pro-inflammatoires conduit au recrutement de cellules immunitaires, en particulier les cellules T auxiliaires CD4+ (TH1), les cellules T cytotoxiques CD8+ et les monocytes, pour monter la défense contre l'infection virale.
Antagoniste de l'IFN de type I, l'Orf6 SARS-CoV-2 inhibe la réponse de l'IFN. L'Orf9b cible le signalosome MAVS pour sa dégradation et limite donc les réponses de l'interféron de la cellule hôte. L'Orf9c interagit avec la chaîne de transport des électrons mitochondriaux qui est impliquée dans la signalisation TLR/IL-1 et la régulation de l'inflammation. Nsp1 supprime l'induction de l'IFN et augmente la production de CCL5, contribuant ainsi aux processus inflammatoires.
SARS-CoV-2 tempête de cytokines & ELISA Kits
Anticorps liés : CRP, NFKB1, NFKB2, NFkB cRel, p65, RELB
- (2)
- (7)
- (1)
- (60)
- (5)
- (2)
- (4)
- (5)
ORF Protéines:
Nos produits sont destinés à la recherche uniquement, et non à un usage diagnostique, thérapeutique ou autre.
Information et produits connexes
SARS-CoV-2 Neutralizing Antibodies based on CR3022
SARS-CoV Protein Interactome / poster and products
SARS-CoV-2 RT-PCR Kits and qPCR Kits
SARS-CoV-2 Life Cycle: Stages and Inhibition Targets
Coronavirus Expression Vectors and Cloning Vectors at genomics-online.com.
Références
- Banerjee, A.K. et al. SARS-CoV-2 Disrupts Splicing, Translation,and Protein Trafficking to Suppress Host Defenses. Cell. doi: 10.1016/j.cell.2020.10.004. Link to Pubmed
- Blanco-Melo, D. et al. Imbalanced Host Response to SARS-CoV-2 Drives Development of COVID-19. Cell. 181, 1036 (2020). Link to Pubmed
- Coutard, B. et al. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Res. 176, 104742 (2020). Link to Pubmed
- Daly, J. et al. Neuropilin-1 is a host factor for SARS-CoV-2 infection. Science (2020). doi: 10.1126/science.abd3072 Link to Pubmed
- de Wilde, A. H. et al. Host Factors in Coronavirus Replication. in Roles of Host Gene and Non-coding RNA Expression in Virus Infection. Current Topics in Microbiology and Immunology (eds. Tripp, R. A. & Tompkins, S. M.) 1–42 (Springer International Publishing, 2018). doi:10.1007/82_2017_25 Link to Pubmed
- Diao, B. et al. Reduction and Functional Exhaustion of T Cells in Patients With Coronavirus Disease 2019 (COVID-19). Front. Immunol. 11, 827 (2020). Link to frontiersin.org
- Gao, T. et al. Highly pathogenic coronavirus N protein aggravates lung injury by MASP-2- mediated complement over-activation. medRxiv (2020). doi:10.1101/2020.03.29.20041962 Link to medRxiv
- Gao, Y. et al. Structure of RNA-dependent RNA polymerase from 2019-nCoV, a major antiviral drug target. bioRxiv 2020.03.16.993386 (2020). doi:10.1101/2020.03.16.993386 Link to bioRxiv
- Gordon, D. E. et al. A SARS-CoV-2 protein interaction map reveals targets for drug repurposing. Nature (2020). doi:10.1038/s41586-020-2286-9 Link to nature.com
- Hoffmann, Markus; Kleine-Weber, H. et al. SARS-CoV-2 Cell Entry Depends on ACE2 and TMPRSS2 and Is Blocked by a Clinically Proven Protease Inhibitor. Cell 181, 1–10 (2020). Link to Pubmed
- Pachetti, M. et al. Emerging SARS‑CoV‑2 mutation hot spots include a novel RNA-dependent- RNA polymerase variant. J. Transl. Med. 18, 1–9 (2020). Link to Pubmed
- Shi, Y. et al. COVID-19 infection: the perspectives on immune responses. Cell Death Differ. (2020). doi:10.1038/s41418-020-0530-3 Link to Pubmed
- Tan, C. et al. C‐reactive protein correlates with computed tomographic findings and predicts severe COVID‐19 early. J. Med. Virol. jmv.25871 (2020). doi:10.1002/jmv.25871 Link to Pubmed
- Tay, M. et al. The trinity of COVID-19: immunity, inflammation and intervention. Nat. Rev. Immunol. 1–12 (2020). doi:10.1038/s41577-020-0311-8 Link to Pubmed
- van den Berg, D. H., te Velde, A. A. Severe COVID-19: NLRP3 Inflammasome Dysregulated. Frontiers in Immunology (2020). doi: 10.3389/fimmu.2020.01580 Link to Pubmed
- Wilk, A. et al. A single-cell atlas of the peripheral immune response in patients with severe COVID-19. Nat.Med. (2020). doi:10.1101/2020.04.17.20069930 Link to Pubmed
- Wrapp, D. et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 367, 1260–1263 (2020). Link to Pubmed
- Yap, J. K. Y. et al. Inflammasomes and Pyroptosis as Therapeutic Targets for COVID-19. Journal of Immunology 205, 307-312(2020). Link to Pubmed
- Zhang, L. et al. Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors. Science. 368, 409 LP – 412 (2020). Link to Pubmed
- Zhang, D. et al. COVID-19 infection induces readily detectable morphological and inflammation-related phenotypic changes in peripheral blood monocytes, the severity of which correlate with patient outcome. medRxiv (2020). doi:10.1101/2020.03.24.20042655 Link to medRxiv
- Ziegler-Heitbrock, L. The CD14+ CD16+ blood monocytes: their role in infection and inflammation. J. Leukoc. Biol. 81, 584–592 (2007). Link to Pubmed




