Les exosomes sont des microvésicules extracellulaires (EMV) excrétées par la plupart des cellules impliquées dans la communication intercellulaire. Ils sont présents dans les fluides corporels et peuvent contenir une vaste gamme de protéines différentes en fonction de leur cellule hôte. Leur contenu est en outre modulé par l'état cellulaire tel que le stress ou l'activation, ou l'inhibition de voies de signalisation spécifiques. Les exosomes sont donc d'excellents biomarqueurs pour les biopsies liquides, par exemple pour le diagnostic du cancer.
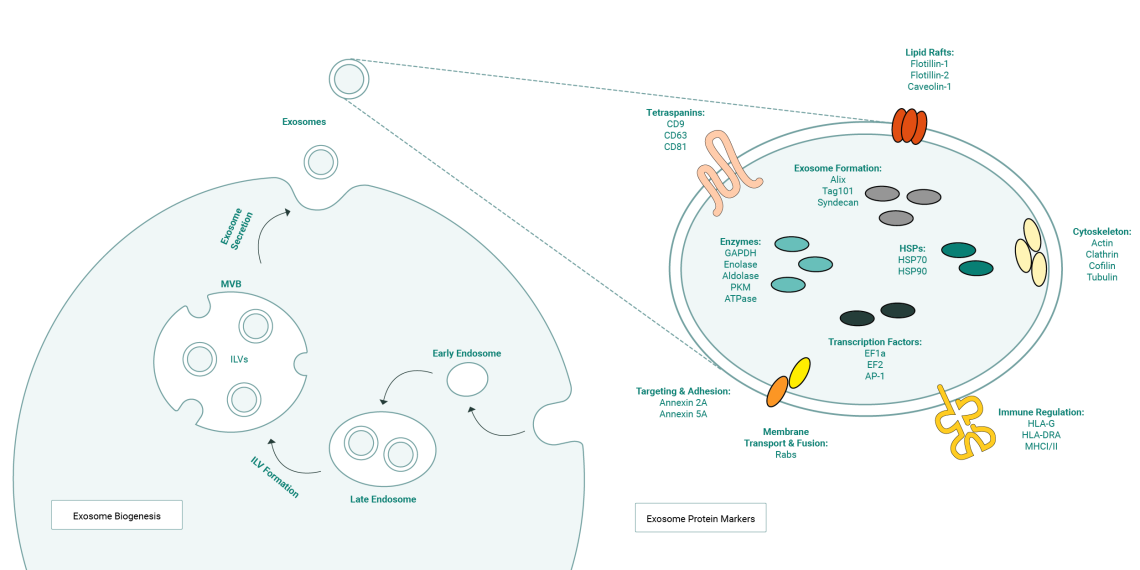
La biogenèse des exosomes commence par l'invagination des endosomes précoces. Ceux-ci se transforment en endosomes tardifs, puis en corps multivésiculaires (MVB). La formation des MVB implique le bourgeonnement vers l'intérieur de la membrane endosomale, ce qui entraîne la séquestration des composants cytoplasmiques dans des vésicules intraluminales (ILV). Les ILV sont libérées par exocytose dans l'espace extracellulaire, ce qui a valu aux exosomes leur nom. Les exosomes peuvent contenir une grande variété de marqueurs protéiques d'exosomes en fonction de leur cellule hôte et de l'état de la cellule (stress, activation ou inhibition de voies de signalisation spécifiques). Les tétraspanines telles que CD9, CD63 et CD81 sont les protéines marqueurs canoniques d'exosomes les plus courantes. La localisation en surface des tétraspanines les rend particulièrement adaptées à l'immunomarquage et à la purification des exosomes à partir d'échantillons biologiques. Les composants du complexe de tri endosomal requis pour le transport (ESCRT) comme TSG101 et Alix, les protéines du cytosquelette, les intégrines et les annexines sont également enrichis dans les exosomes. Ces molécules jouent un rôle essentiel dans le ciblage des exosomes et l'adhésion cellulaire.
Que sont les exosomes ?
Les exosomes sont de petites microvésicules extracellulaires (EMV) dérivées des endosomes (50-120 nm) qui jouent un rôle crucial dans la communication intercellulaire en transportant diverses biomolécules, telles que des protéines, des lipides et des acides nucléiques, entre les cellules. Elles ont été observées pour la première fois au début des années 1980 dans les milieux de culture des réticulocytes. Le terme exosome est basé sur l'observation qu'ils sont libérés par exocytose dans le milieu extracellulaire. Les exosomes ont une topologie similaire à celle de la membrane plasmique et sont libérés par pratiquement tous les types de cellules ; leur présence a été confirmée dans tous les fluides corporels.
Téléchargez notre poster sur les marqueurs d'exosomes !
En cliquant sur le lien ci-dessous, vous pouvez télécharger une copie de notre poster sur les exosomes au format PDF.
Demander une copieComment se forment les exosomes ?
La biogenèse des exosomes commence généralement par l'internalisation du matériel extracellulaire par endocytose, ce qui conduit à la formation d'endosomes précoces, des organites membranaires qui contiennent la cargaison internalisée. Ces endosomes deviennent ensuite des endosomes tardifs, un processus qui s'accompagne de changements dans la composition de la membrane et dans le tri de la cargaison. Les endosomes tardifs peuvent se transformer en MVB, qui se caractérisent par la présence de vésicules intraluminales (ILV) formées par l'invagination de la membrane endosomale. Dans les MVB, les protéines, les lipides, les acides nucléiques et d'autres molécules sont triés dans les ILV. Ce tri est une étape critique de la biogenèse des exosomes et est réalisé par les complexes de tri endosomal requis pour le transport (ESCRT). L'ESCRT est constitué de plusieurs complexes protéiques (ESCRT-0, ESCRT-I, ESCRT-II, ESCRT-III) qui travaillent ensemble pour reconnaître et séquestrer la cargaison dans les ILV en formation. Les MVB peuvent ensuite fusionner avec les lysosomes pour être dégradés ou avec la membrane plasmique, libérant les ILV dans l'espace extracellulaire sous forme d'exosomes.
Comment les exosomes sont-ils identifiés ?
Les exosomes contiennent une vaste gamme de protéines différentes en fonction de leur cellule hôte, et leurs composants sont encore modulés par l'état cellulaire (par exemple, le stress ou l'activation, ou l'inhibition de voies de signalisation spécifiques). Les tétraspanines comme CD9, CD63 et CD81 sont les protéines marqueurs canoniques des exosomes les plus courantes, présentes à la surface des vésicules. La localisation en surface des antigènes des tétraspanines en fait de bonnes cibles candidates pour l'immunomarquage et la purification des exosomes à partir d'échantillons biologiques. Les composants du complexe de tri endosomal requis pour le transport (ESCRT) comme TSG101 et Alix, les protéines du cytosquelette, les intégrines et les annexines sont également enrichis sur les exosomes ; ces molécules jouent un rôle central dans le ciblage des exosomes et l'adhésion cellulaire.
Pourquoi les exosomes sont-ils importants ?
La sécrétion des exosomes est constitutive, bien que le taux de sécrétion et la composition des exosomes puissent être augmentés par une variété de facteurs intrinsèques ou extrinsèques (par exemple, le stress cellulaire, les cascades de signalisation). Malgré leur nature ubiquitaire, les exosomes sont considérés comme des composants non conventionnels de la voie sécrétoire.
Comme les exosomes sont sécrétés par presque tous les types de cellules, leur composition reflète la diversité de leurs hôtes et dépend fortement du type de cellule dont ils proviennent. Leur composition moléculaire reflète également les changements physiologiques ou pathophysiologiques dans leur cellule ou tissu d'origine. Les exosomes sont donc d'excellents biomarqueurs pour les biopsies liquides, qui permettent de diagnostiquer et de suivre la progression d'une maladie. Pour le diagnostic du cancer, les exosomes présentent des avantages significatifs par rapport aux cellules tumorales circulantes (CTC) ou à l'ADN tumoral circulant (ADNtc) en raison de leur abondance, de leur stabilité et de la grande variété de molécules marqueurs qu'ils contiennent.
Les exosomes sont également impliqués dans la communication entre les cellules. Les composants des exosomes peuvent être transférés directement aux cellules voisines ou peuvent être transportés à travers différentes cellules avant d'atteindre leur destination finale par une méthode connue sous le nom de transcytose. De cette manière, les exosomes peuvent transmettre des signaux sur de grandes distances où la simple diffusion peut être insuffisante. Leur rôle dans la communication cellule-cellule suggère que les exosomes peuvent avoir un rôle plus profond dans de nombreux processus physiologiques ; cette hypothèse est soutenue par l'observation que la signalisation des exosomes joue un rôle direct dans le développement et le modelage, la réponse immunitaire, la communication neuronale et la réparation des tissus.
Dans certaines pathologies, les exosomes peuvent également jouer un rôle dans le développement et le modelage, la réponse immunitaire, la communication neuronale et la réparation des tissus. Dans certaines pathologies, les exosomes agissent également comme des vecteurs ; les exosomes dérivés des cellules tumorales jouent un rôle actif dans l'angiogenèse et la métastase des tumeurs. Les exosomes provenant des cellules sanguines stimulées et de l'endothélium vasculaire sont impliqués dans des troubles neurologiques tels que la sclérose en plaques, les accidents ischémiques transitoires et le syndrome des antiphospholipides. Les exosomes peuvent également transporter du matériel cellulaire endommagé destiné à être détruit et faciliter la propagation de formes toxiques de protéines agrégées telles que l'α-synucléine et la β-amyloïde, et contribuer à la progression des maladies neurodégénératives. Certaines recherches suggèrent également que le transport des exosomes a été exploité par des pathogènes viraux tels que le SARS-CoV-2 pour se déplacer entre les cellules hôtes et échapper à la détection immunitaire. En raison de leur petite taille et de leur structure simple, les exosomes peuvent parfois traverser la barrière hémato-encéphalique. Il a été suggéré que les exosomes pourraient servir de système d'administration ciblant le système nerveux central pour traiter les maladies neuropathiques sans avoir recours à une chirurgie invasive. L'utilisation des exosomes pour transférer des informations génétiques ou pour délivrer des agents thérapeutiques est un domaine actuellement sous-exploré qui recèle un vaste potentiel médical.
Comment les exosomes sont-ils étudiés ?
Le sécrétome des exosomes est vaste et diversifié et contient de nombreux marqueurs différents (voir http://www.exocarta.org/). La présence de marqueurs de surface canoniques tels que ceux énumérés ci-dessus permet la purification et l'étude approfondie de la sécrétion et du contenu des exosomes à partir de différents types d'échantillons.
antibodies-online propose une gamme d'anticorps et de kits ELISA pour la détection des protéines d'exosomes connus.
Anticorps marqueurs d'exosomes
D'après la littérature récente, les marqueurs d'exosomes protéiques les plus pertinents sont les suivants:
| Protein | Gene | GeneID | Uniprot | Ref | exocarta Top 100 proteins | TS | EF | LP | TA | CS | AG | MT | AP | HS | EN | RG | CA | II | VI | ND |
| 14-3-3 protein epsilon | YWHAE | 7531 | P62258 | - | 22 | X | ||||||||||||||
| 14-3-3 protein beta/alpha | YWHAB | 7529 | P31946 | 50 | x | |||||||||||||||
| 14-3-3 protein eta | YWHAH | 7533 | Q04917 | 94 | x | |||||||||||||||
| 14-3-3 protein gamma | YWHAG | 7532 | P61981 | 54 | x | |||||||||||||||
| 14-3-3 protein theta | YWHAQ | 10971 | P27348 | 56 | x | |||||||||||||||
| 14-3-3 protein zeta/delta | YWHAZ | 7534 | P63104 | - | 15 | X | ||||||||||||||
| 78 kDa glucose-regulated protein | HSPA5 | 3309 | P11021 | (1) | 35 | X | ||||||||||||||
| Actin, cytoplasmic 1 | ACTB | 60 | P60709 | - | 5 | X | ||||||||||||||
| ADAM10 | ADAM10 | 102 | O14672 | (2) | - | X | X | X | X | |||||||||||
| Alix | PDCD6IP | 10015 | Q8WUM4 | (3) | 2 | X | ||||||||||||||
| Alpha-Enolase | ENO1 | 2023 | P06733 | (4) | 9 | X | ||||||||||||||
| Alpha-Synclein | SNCA | 6622 | P37840 | (32) | - | X | X | |||||||||||||
| Aminopeptidase N | ANPEP | 290 | P15144 | - | - | X | X | |||||||||||||
| Beta-amyloid | APP | 351 | P05067 | (5) | - | X | ||||||||||||||
| Annexin A1 | ANXA1 | 301 | P04083 | 53 | x | |||||||||||||||
| Annexin A11 | ANXA11 | 311 | P50995 | 68 | x | |||||||||||||||
| Annexin A4 | ANXA4 | 307 | P09525 | 72 | x | |||||||||||||||
| Annexin A6 | ANXA6 | 309 | P08133 | 67 | x | |||||||||||||||
| Annexin A5 | ANXA5 | 308 | P08758 | (6) | 20 | X | X | |||||||||||||
| Annexin A2 | ANXA2 | 302 | P07355 | (7) | 6 | X | ||||||||||||||
| AP-1 | JUN | 3725 | P05412 | - | - | X | X | |||||||||||||
| ATP citrate lyase | ACLY | 47 | P53396 | - | 72 | X | ||||||||||||||
| ATPase | ATP1A1 | 476 | P05023 | - | 39 | X | ||||||||||||||
| Basigin | BSG | 682 | P35613 | (8) | - | X | X | |||||||||||||
| Caveolin-1 | CAV1 | 857 | Q03135 | (9), (10) | - | X | X | |||||||||||||
| CD9 | CD9 | 928 | P21926 | (11) | 1 | X | ||||||||||||||
| CD11a | ITGAL | 3683 | P20701 | (12) | - | X | X | |||||||||||||
| CD11b | ITGAX | 3687 | P11215 | (12) | - | X | X | |||||||||||||
| CD11c | ITGAM | 3684 | P20702 | (12) | - | X | X | |||||||||||||
| CD29 | ITGB1 | 3688 | P05556 | (12) | 34 | X | X | |||||||||||||
| CD37 | CD37 | 951 | P11049 | (11) | - | X | ||||||||||||||
| CD44 | CD44 | 960 | P16070 | (13) | - | X | X | |||||||||||||
| CD49f | ITGA6 | 3655 | P23229 | (12) | 89 | X | X | |||||||||||||
| CD53 | CD53 | 963 | P19397 | - | x | |||||||||||||||
| CD63 | CD63 | 967 | P08962 | (10), (11) | 7 | X | X | |||||||||||||
| CD81 | CD81 | 975 | P60033 | (14) | 24 | X | X | |||||||||||||
| CD82 | CD82 | 3732 | P27701 | (11) | - | X | ||||||||||||||
| CD142 | TF | 2152 | P13726 | (15) | - | X | X | |||||||||||||
| CD146 | MCAM | 4162 | P43121 | (15) | - | X | X | |||||||||||||
| CD163 | CD163 | 9332 | Q86VB7 | (15) | - | X | X | X | ||||||||||||
| Clathrin heavy chain 1 | CLTC | 1213 | Q00610 | - | 23 | X | ||||||||||||||
| Claudin-1 | CLDN1 | 9076 | O95832 | (8) | - | X | ||||||||||||||
| Cofilin-1 | CFL1 | 1072 | P23528 | - | 25 | X | ||||||||||||||
| - | - | (16) | - | X | ||||||||||||||||
| - | - | (16) | - | X | ||||||||||||||||
| EF-1-alpha-1 | EEF1A1 | 1915 | P68104 | (4) | 14 | X | ||||||||||||||
| EF2 | EEF2 | 1938 | P13639 | - | 17 | X | ||||||||||||||
| EGFR | EGFR | 1956 | P00533 | (15) | - | X | ||||||||||||||
| Ep-CAM | EPCAM | 4072 | P16422 | (17), (18) | - | X | X | |||||||||||||
| Fatty acid synthase | FASN | 2194 | P49327 | (3) | 21 | X | X | |||||||||||||
| Fibronectin | FN1 | 2335 | P02751 | 93 | ||||||||||||||||
| Flotillin-1 | FLOT1 | 10211 | O75955 | (18), (19) | 41 | X | X | |||||||||||||
| Flotillin-2 | FLOT2 | 2319 | Q14254 | (19) | - | X | ||||||||||||||
| Fructose-bisphosphate aldolase A | ALDOA | 226 | P04075 | - | 18 | X | ||||||||||||||
| Gelsolin | GSN | 2934 | P06396 | 92 | x | x | ||||||||||||||
| Glyceraldehyde-3-phosphate dehydrogenase | GAPDH | 2597 | P04406 | - | 4 | X | ||||||||||||||
| HCV core protein | - | - | (20) | - | X | |||||||||||||||
| Heat shock 70 kDa protein 1A | HSPA1A | 3303 | P0DMV8 | 51 | x | |||||||||||||||
| Heat shock protein HSP 90-alpha | HSP90AA1 | 3320 | P07900 | (1) | 10 | X | X | |||||||||||||
| Heat shock protein HSP 90-beta | HSP90AB1 | 3326 | P08238 | (1) | 19 | X | ||||||||||||||
| Heparanase | HPSE | 10855 | Q9Y251 | (21) | - | X | X | |||||||||||||
| - | - | (20) | - | X | ||||||||||||||||
| - | - | (20) | - | X | ||||||||||||||||
| HLA-DRA | HLA-DRA | 3122 | P01903 | (22) | - | X | X | X | ||||||||||||
| HLA-G | 3135 | P17693 | (23) | - | X | X | X | |||||||||||||
| Hsc70 | HSPA8 | 3312 | P11142 | (1) | 3 | X | ||||||||||||||
| - | - | (16) | - | X | ||||||||||||||||
| Tax | - | - | (20) | - | X | |||||||||||||||
| Huntingtin | HTT | 3064 | P42858 | (5) | - | X | X | |||||||||||||
| ICAM-1 | ICAM1 | 3383 | P05362 | (24) | - | X | ||||||||||||||
| Leucine-rich receptor kinase 2 | LRRK2 | 120892 | Q5S007 | (5) | - | X | ||||||||||||||
| L-lactate dehydrogenase A chain | LDHA | 3939 | P00338 | - | 13 | X | ||||||||||||||
| Lysosome-associated membrane glycoprotein 1 | LAMP1 | 3916 | P11279 | (25) | - | X | X | |||||||||||||
| Lysosome-associated membrane glycoprotein 2 | LAMP2 | 3920 | P13473 | (23) | 88 | X | X | |||||||||||||
| MHCI | - | - | (26) | - | X | X | ||||||||||||||
| MHCII | - | - | (26) | - | X | X | ||||||||||||||
| MUC1 | MUC1 | 4582 | P15941 | (15) | - | X | X | |||||||||||||
| N-cadherin | CDH2 | 1000 | P19022 | (15) | - | X | X | |||||||||||||
| Phosphoglycerate kinase 1 | PGK1 | 5230 | P00558 | (4) | 16 | X | ||||||||||||||
| Placental Alkaline Phosphatase | ALPP | 250 | P05187 | (15) | - | X | X | |||||||||||||
| Prion proteins | - | - | (5) | - | X | |||||||||||||||
| Prostate-specific antigen | KLK3 | 354 | P07288 | (27) | - | X | X | |||||||||||||
| Pyruvate kinase PKM | PKM | 5315 | P14618 | (28) | 12 | X | X | |||||||||||||
| Rab-14 | RAB14 | 51552 | P61106 | - | 75 | X | ||||||||||||||
| Rab-5a | RAB5A | 5868 | P20339 | - | 80 | X | ||||||||||||||
| Rab-5b | RAB5B | 5869 | P61020 | - | 86 | X | ||||||||||||||
| Rab-5c | RAB5C | 5878 | P51148 | - | 64 | X | ||||||||||||||
| Rab-7 | RAB7A | 7879 | P51149 | - | 61 | X | ||||||||||||||
| Rap 1B | RAP1B | 5908 | P61224 | - | 33 | X | ||||||||||||||
| Superoxide dismutase | SOD1 | 6647 | P00441 | - | x | x | ||||||||||||||
| Syndecan-1 | SDC1 | 6382 | P18827 | (29) | - | X | ||||||||||||||
| Syndecan-4 | SDC4 | 6385 | P31431 | (29) | - | X | ||||||||||||||
| Syntenin-1 | SDCBP | 6386 | O00560 | (30) | 8 | X | ||||||||||||||
| TARDBP | TDP-43 | 23435 | Q13148 | - | x | |||||||||||||||
| Transitional endoplasmic reticulum ATPase | VCP | 7415 | P55072 | 26 | ||||||||||||||||
| Triosephosphate isomerase | TPI1 | 7167 | P60174 | 27 | x | |||||||||||||||
| Tumor-Associated Glycoprotein | TAG-72 | - | - | (15) | - | X | ||||||||||||||
| Tetraspanin-8 | Tspan8 | 7103 | P19075 | (15) | - | X | X | |||||||||||||
| Tsg101 | TSG101 | 7251 | Q99816 | (31) | 11 | X | ||||||||||||||
| Tubulin alpha-1C chain | TUBA1C | 84790 | Q9BQE3 | - | x | |||||||||||||||
| Tubulin alpha-4A chain | TUBA4A | 7277 | P68366 | - | x | |||||||||||||||
| Tubulin beta-2B chain | TUBB2B | 347733 | Q9BVA1 | - | x | |||||||||||||||
| Tubulin beta-4B chain | TUBB4B | 10383 | P68371 | - | x | |||||||||||||||
| Vacuolar-sorting protein 35 | VPS35 | 55737 | Q96QK1 | (5) | - | X | X |

Téléchargez notre poster sur les marqueurs d'exosomes !
En cliquant sur le lien ci-dessous, vous pouvez télécharger une copie de notre poster sur les exosomes au format PDF.
Demander une copieRéférences
- : "ICAM-1 on exosomes from mature dendritic cells is critical for efficient naive T-cell priming." dans: Blood, Vol. 106, Issue 1, pp. 216-23, (2005) (PubMed).
- : "Exosome-dependent trafficking of HSP70: a novel secretory pathway for cellular stress proteins." dans: The Journal of biological chemistry, Vol. 280, Issue 24, pp. 23349-55, (2005) (PubMed).
- : "The regulation of exosome secretion: a novel function of the p53 protein." dans: Cancer research, Vol. 66, Issue 9, pp. 4795-801, (2006) (PubMed).
- : "B cell activation regulates exosomal HLA production." dans: European journal of immunology, Vol. 38, Issue 5, pp. 1423-34, (2008) (PubMed).
- : "CD44 and EpCAM: cancer-initiating cell markers." dans: Current molecular medicine, Vol. 8, Issue 8, pp. 784-804, (2009) (PubMed).
- : "High levels of exosomes expressing CD63 and caveolin-1 in plasma of melanoma patients." dans: PloS one, Vol. 4, Issue 4, pp. e5219, (2009) (PubMed).
- : "Claudin-containing exosomes in the peripheral circulation of women with ovarian cancer." dans: BMC cancer, Vol. 9, pp. 244, (2009) (PubMed).
- : "Lipid raft endocytosis and exosomal transport facilitate extracellular trafficking of annexin A2." dans: The Journal of biological chemistry, Vol. 286, Issue 35, pp. 30911-30925, (2011) (PubMed).
- : "Microvesicles and viral infection." dans: Journal of virology, Vol. 85, Issue 24, pp. 12844-54, (2012) (PubMed).
- : "The roles of flotillin microdomains--endocytosis and beyond." dans: Journal of cell science, Vol. 124, Issue Pt 23, pp. 3933-40, (2012) (PubMed).
- : "Soluble serum CD81 is elevated in patients with chronic hepatitis C and correlates with alanine aminotransferase serum activity." dans: PloS one, Vol. 7, Issue 2, pp. e30796, (2012) (PubMed).
- : "Syndecan-syntenin-ALIX regulates the biogenesis of exosomes." dans: Nature cell biology, Vol. 14, Issue 7, pp. 677-85, (2012) (PubMed).
- : "Quantitative proteomic analysis of exosomes from HIV-1-infected lymphocytic cells." dans: Proteomics, Vol. 12, Issue 13, pp. 2203-11, (2012) (PubMed).
- : "Exosomes and microvesicles: extracellular vesicles for genetic information transfer and gene therapy." dans: Human molecular genetics, Vol. 21, Issue R1, pp. R125-34, (2013) (PubMed).
- : "Innate immune response of human alveolar type II cells infected with severe acute respiratory syndrome-coronavirus." dans: American journal of respiratory cell and molecular biology, Vol. 48, Issue 6, pp. 742-8, (2013) (PubMed).
- : "Heparanase regulates secretion, composition, and function of tumor cell-derived exosomes." dans: The Journal of biological chemistry, Vol. 288, Issue 14, pp. 10093-10099, (2013) (PubMed).
- : "Exosome uptake depends on ERK1/2-heat shock protein 27 signaling and lipid Raft-mediated endocytosis negatively regulated by caveolin-1." dans: The Journal of biological chemistry, Vol. 288, Issue 24, pp. 17713-24, (2013) (PubMed).
- : "Proteomic profiling of exosomes leads to the identification of novel biomarkers for prostate cancer." dans: PloS one, Vol. 8, Issue 12, pp. e82589, (2014) (PubMed).
- : "Syntenin-ALIX exosome biogenesis and budding into multivesicular bodies are controlled by ARF6 and PLD2." dans: Nature communications, Vol. 5, pp. 3477, (2014) (PubMed).
- : "Plasma exosomal α-synuclein is likely CNS-derived and increased in Parkinson's disease." dans: Acta neuropathologica, Vol. 128, Issue 5, pp. 639-650, (2015) (PubMed).
- : "Tetraspanins in extracellular vesicle formation and function." dans: Frontiers in immunology, Vol. 5, pp. 442, (2014) (PubMed).
- : "Exosomes secreted by cortical neurons upon glutamatergic synapse activation specifically interact with neurons." dans: Journal of extracellular vesicles, Vol. 3, pp. 24722, (2014) (PubMed).
- : "Antigen Presentation by MHC-Dressed Cells." dans: Frontiers in immunology, Vol. 5, pp. 672, (2015) (PubMed).
- : "Anti-cancer fatty-acid derivative induces autophagic cell death through modulation of PKM isoform expression profile mediated by bcr-abl in chronic myeloid leukemia." dans: Cancer letters, Vol. 360, Issue 1, pp. 28-38, (2015) (PubMed).
- : "Exosomes: novel biomarkers for clinical diagnosis." dans: TheScientificWorldJournal, Vol. 2015, pp. 657086, (2016) (PubMed).
- : "Exosomal proteins as potential diagnostic markers in advanced non-small cell lung carcinoma." dans: Journal of extracellular vesicles, Vol. 4, pp. 26659, (2015) (PubMed).
- : "HLA-G: An Immune Checkpoint Molecule." dans: Advances in immunology, Vol. 127, pp. 33-144, (2015) (PubMed).
- : "Exosomes and Their Role in the Life Cycle and Pathogenesis of RNA Viruses." dans: Viruses, Vol. 7, Issue 6, pp. 3204-25, (2016) (PubMed).
- : "ExoCarta: A Web-Based Compendium of Exosomal Cargo." dans: Journal of molecular biology, Vol. 428, Issue 4, pp. 688-692, (2016) (PubMed).
- : "Tumour exosome integrins determine organotropic metastasis. ..." dans: Nature, Vol. 527, Issue 7578, pp. 329-35, (2015) (PubMed).
- : "Exosomes in neurological disease, neuroprotection, repair and therapeutics: problems and perspectives." dans: Neural regeneration research, Vol. 10, Issue 10, pp. 1565-7, (2015) (PubMed).
- : "Interrogating Circulating Microsomes and Exosomes Using Metal Nanoparticles." dans: Small (Weinheim an der Bergstrasse, Germany), Vol. 12, Issue 6, pp. 727-32, (2016) (PubMed).
- : "PSA and beyond: alternative prostate cancer biomarkers." dans: Cellular oncology (Dordrecht), Vol. 39, Issue 2, pp. 97-106, (2016) (PubMed).
- : "A Perspective on Extracellular Vesicles Proteomics." dans: Frontiers in chemistry, Vol. 5, pp. 102, (2017) (PubMed).
- : "Reassessment of Exosome Composition." dans: Cell, Vol. 177, Issue 2, pp. 428-445.e18, (2019) (PubMed).
- : "On the potential role of exosomes in the COVID-19 reinfection/reactivation opportunity." dans: Journal of biomolecular structure & dynamics, Vol. 39, Issue 15, pp. 5831-5842, (2021) (PubMed).
- : "Ultrastructural Evidence for Direct Renal Infection with SARS-CoV-2." dans: Journal of the American Society of Nephrology : JASN, Vol. 31, Issue 8, pp. 1683-1687, (2020) (PubMed).
- : "Extracellular vesicles and amyotrophic lateral sclerosis: from misfolded protein vehicles to promising clinical biomarkers." dans: Cellular and molecular life sciences : CMLS, Vol. 78, Issue 2, pp. 561-572, (2021) (PubMed).
- : "The exosome journey: from biogenesis to uptake and intracellular signalling." dans: Cell communication and signaling : CCS, Vol. 19, Issue 1, pp. 47, (2022) (PubMed).
- : "Emerging Role of Exosomes in Liquid Biopsy for Monitoring Prostate Cancer Invasion and Metastasis." dans: Frontiers in cell and developmental biology, Vol. 9, pp. 679527, (2021) (PubMed).
- : "Valosin-Containing Protein (VCP)/p97: A Prognostic Biomarker and Therapeutic Target in Cancer." dans: International journal of molecular sciences, Vol. 22, Issue 18, (2021) (PubMed).
- : "Exosomes as a new frontier of cancer liquid biopsy." dans: Molecular cancer, Vol. 21, Issue 1, pp. 56, (2022) (PubMed).
- : "Gelsolin: A comprehensive pan-cancer analysis of potential prognosis, diagnostic, and immune biomarkers." dans: Frontiers in genetics, Vol. 14, pp. 1093163, (2023) (PubMed).
- : "Vesiclepedia 2024: an extracellular vesicles and extracellular particles repository." dans: Nucleic acids research, Vol. 52, Issue D1, pp. D1694-D1698, (2024) (PubMed).
- : "Roles of exosomes in immunotherapy for solid cancers." dans: Cell death & disease, Vol. 15, Issue 2, pp. 106, (2024) (PubMed).





